Sebuah haplotype adalah wilayah genom yang cenderung diwariskan bersama-sama melalui beberapa generasi; biasanya itu semua terletak pada kromosom yang sama. Haplotipe adalah produk dari hubungan genetik dan tetap utuh selama rekombinasi genetik.
Kata “haplotipe” berasal dari gabungan kata “haploid” dan kata “genotipe”. “Haploid” mengacu pada sel dengan satu set kromosom dan “genotipe” mengacu pada susunan genetik suatu organisme.
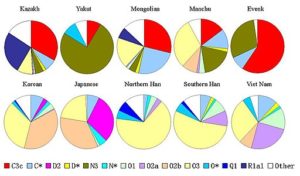
Skema distribusi haplotipe kromosom Y pada populasi Asia (Sumber: Moogalord [Domain publik] melalui Wikimedia Commons)
Mengikuti definisi, haplotipe dapat menggambarkan sepasang gen atau lebih yang diwarisi bersama pada kromosom dari orang tua, atau dapat menggambarkan kromosom yang diwarisi seluruhnya dari orang tua, seperti halnya kromosom Y pada pria. .
Misalnya, ketika haplotipe berbagi gen untuk dua sifat fenotipik yang berbeda, seperti warna rambut dan warna mata, individu yang memiliki gen untuk warna rambut juga akan memiliki gen lain untuk warna mata.
Haplotipe adalah salah satu alat yang paling banyak digunakan saat ini untuk studi silsilah, untuk melacak asal usul penyakit, untuk mengkarakterisasi variabilitas genetik dan filgeografi populasi berbagai jenis makhluk hidup.
Ada beberapa alat untuk mempelajari haplotipe, salah satu yang paling banyak digunakan saat ini adalah ” Peta haplotipe ” (HapMap), yang merupakan halaman web yang memungkinkan untuk menentukan segmen genom mana yang merupakan haplotipe.
Indeks artikel
Metode belajar
Haplotipe mewakili kesempatan untuk memahami pewarisan gen dan polimorfismenya. Dengan penemuan seni “reaksi berantai polimerase” (PCR, dari bahasa Inggris ” Reaksi Berantai Polimerase “), itu dikembangkan secara luas dalam studi haplotipe.
Saat ini ada banyak metodologi untuk mempelajari haplotipe, beberapa yang paling menonjol adalah:
Sekuensing DNA dan deteksi polimorfisme nukleotida tunggal (SNPs)
Perkembangan teknologi sekuensing generasi berikutnya merupakan lompatan besar untuk studi haplotipe. Teknologi baru memungkinkan untuk mendeteksi variasi hingga satu basa nukleotida di wilayah tertentu dari suatu haplotipe.
Dalam bioinformatika, istilah haplotipe juga digunakan untuk merujuk pada pewarisan sekelompok polimorfisme nukleotida tunggal (SNP) dalam urutan DNA .
Dengan menggabungkan program bioinformatika dengan deteksi haplotipe menggunakan sekuensing generasi berikutnya, posisi, substitusi, dan efek dari setiap perubahan basa dalam genom suatu populasi dapat diidentifikasi secara akurat.
Mikrosatelit (SSRS)
Mikrosatelit atau SSRS, mendapatkan namanya dari bahasa Inggris “S imple Sequence Repeat and Short Tandem Repeat ”. Ini adalah urutan nukleotida pendek yang diulang secara berurutan dalam suatu wilayah genom.
Adalah umum untuk menemukan mikrosatelit di dalam haplotipe non-coding, oleh karena itu, melalui deteksi variasi jumlah pengulangan mikrosatelit, alel yang berbeda dalam haplotipe individu dapat diamati.
Penanda mikrosatelit molekuler telah dikembangkan untuk mendeteksi berbagai haplotipe, mulai dari jenis kelamin tanaman seperti Pepaya ( Carica papaya ) hingga deteksi penyakit manusia seperti anemia sel sabit.
Polimorfisme panjang fragmen yang diperkuat (AFLP)
Teknik ini menggabungkan amplifikasi dengan reaksi PCR dengan pencernaan DNA dengan dua enzim restriksi yang berbeda. Teknik ini mendeteksi lokus polimorfik dalam haplotipe menurut situs pembelahan yang berbeda dalam urutan DNA.
Untuk mengilustrasikan teknik dengan lebih baik, mari kita bayangkan tiga fragmen kain dengan panjang yang sama, tetapi dipotong di tempat yang berbeda (fragmen ini mewakili tiga fragmen haplotipe yang diperkuat PCR).
Pada saat kain dipotong, akan diperoleh banyak potongan dengan ukuran yang berbeda, karena setiap kain dipotong di tempat yang berbeda. Dengan mengurutkan fragmen menurut jenis kain asalnya, kita akan dapat mengamati di mana perbedaan antara kain atau haplotipe ditemukan.
Diagnosa dan penyakit
Keuntungan penting dari studi genetik haplotipe adalah bahwa mereka tetap hampir utuh atau tidak berubah selama ribuan generasi, dan ini memungkinkan identifikasi nenek moyang yang jauh dan setiap mutasi yang berkontribusi pada perkembangan penyakit.
Haplotipe pada manusia bervariasi tergantung pada ras dan, berdasarkan yang pertama, gen telah terdeteksi di dalam haplotipe yang menyebabkan penyakit parah pada setiap ras manusia.
Empat kelompok ras termasuk dalam proyek HapMap : Eropa, Nigeria, Yoruba, Han Cina, dan Jepang.
Dengan cara ini, proyek HapMap dapat mencakup kelompok populasi yang berbeda dan melacak asal usul dan evolusi banyak penyakit bawaan yang mempengaruhi masing-masing dari empat breed.
Salah satu penyakit yang paling sering didiagnosis menggunakan analisis haplotipe adalah anemia sel sabit pada manusia. Penyakit ini didiagnosis dengan melacak frekuensi haplotipe Afrika dalam suatu populasi.
Menjadi penyakit asli Afrika, mengidentifikasi haplotipe Afrika dalam populasi memudahkan untuk melacak orang yang memiliki mutasi dalam urutan genetik untuk globin beta dalam eritrosit berbentuk sabit (karakteristik penyakit).
Contoh
Dengan haplotipe, pohon filogenetik dibangun yang mewakili hubungan evolusioner antara masing-masing haplotipe yang ditemukan dalam sampel molekul DNA homolog atau spesies yang sama, di wilayah yang memiliki sedikit atau tanpa rekombinasi.
Salah satu cabang yang paling banyak dipelajari melalui haplotipe adalah evolusi sistem kekebalan manusia. Haplotipe yang mengkode reseptor seperti TOll (komponen kunci dari sistem kekebalan bawaan) telah diidentifikasi untuk genom Neanderthal dan Denisovan.
Ini memungkinkan mereka untuk melacak bagaimana urutan genetik dalam populasi manusia “cararn” telah berubah dari urutan haplotipe yang sesuai dengan manusia “leluhur”.
Dengan membangun jaringan hubungan genetik dari haplotipe mitokondria, dipelajari bagaimana efek pendiri terjadi pada spesies, karena ini memungkinkan para ilmuwan untuk mengidentifikasi kapan populasi berhenti bereproduksi di antara mereka sendiri dan menetapkan diri mereka sebagai spesies terpisah.
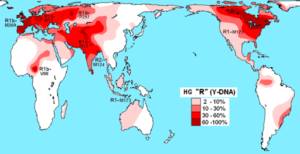
Distribusi haplogroup R (DNA-Y) pada populasi asli (Sumber: Maulucioni [CC BY 3.0 (https://creativecommons.org/licenses/by/3.0)], melalui Wikimedia Commons)
Keragaman haplotipe digunakan untuk melacak dan mempelajari keragaman genetik hewan penangkaran. Teknik ini digunakan terutama untuk spesies yang sulit dipantau di alam liar.
Spesies hewan seperti hiu, burung, dan mamalia besar seperti jaguar, gajah, antara lain, secara terus-menerus dievaluasi secara genetik melalui haplotipe mitokondria untuk memantau status genetik populasi di penangkaran.
Referensi
- Bahlo, M., Stankovich, J., Kecepatan, TP, Rubio, JP, Burfoot, RK, & Foote, SJ (2006). Mendeteksi berbagi haplotipe luas genom menggunakan data haplotipe SNP atau mikrosatelit. Genetika manusia, 119 (1-2), 38-50.
- Dannemann, M., Andrés, AM, & Kelso, J. (2016). Introgresi haplotipe mirip Neandertal dan Denisovan berkontribusi pada variasi adaptif pada reseptor mirip Tol manusia. American Journal of Human Genetics, 98 (1), 22-33.
- De Vries, HG, van der Meulen, MA, Rozen, R., Halley, DJ, Scheffer, H., Leo, P., … & te Meerman, GJ (1996). Identitas haplotipe antara individu yang berbagi alel mutasi CFTR “identik dengan keturunan”: demonstrasi kegunaan konsep berbagi haplotipe untuk pemetaan gen dalam populasi nyata. Genetika manusia, 98 (3), 304-309
- Degli-Esposti, MA, Leaver, AL, Christiansen, FT, Witt, CS, Abraham, LJ, & Dawkins, RL (1992). Haplotipe leluhur: haplotipe MHC populasi yang dilestarikan. Imunologi manusia, 34 (4), 242-252.
- Fellows, MR, Hartman, T., Hermelin, D., Landau, GM, Rosamond, F., & Rozenberg, L. (2009, Juni). Inferensi haplotipe dibatasi oleh data haplotipe yang masuk akal. Dalam Simposium Tahunan tentang Pencocokan Pola Kombinatorial (hlm. 339-352). Springer, Berlin, Heidelberg.
- Gabriel, SB, Schaffner, SF, Nguyen, H., Moore, JM, Roy, J., Blumenstiel, B., … & Liu-Cordero, SN (2002). Struktur blok haplotype dalam genom manusia. Sains, 296 (5576), 2225-2229.
- Konsorsium HapMap Internasional. (2005). Peta haplotipe genom manusia. Alam, 437 (7063), 1299.
- Wynne, R., & Wilding, C. (2018). Keanekaragaman haplotipe DNA mitokondria dan asal penangkaran hiu macan pasir (Carcharias taurus). Jurnal Penelitian Kebun Binatang dan Akuarium, 6 (3), 74-78.
- Yoo, YJ, Tang, J., Kaslow, RA, & Zhang, K. (2007). Inferensi haplotipe untuk data genotipe saat ini – tidak ada menggunakan haplotipe dan pola haplotipe yang diidentifikasi sebelumnya. Bioinformatika, 23 (18), 2399-2406.
- Muda, NS (2018). Anemia aplastik. New England Journal of Medicine, 379 (17), 1643-1656.

